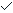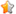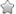对差异表达基因表达谱数据进行深度挖掘,看如何做PPI(蛋白-蛋白相互作用)生信分析发个2-3分的文章。
文章标题及来源如下:

这篇文章作者利用他们之前已经做了的食管癌细胞敲除Ezrin基因后得到差异表达基因(DEGs)数据,只做PPI生信分析的发在了BioMed Research International(IF:2.5)。
首先是PPI网络的构建:
作者用了人实验验证的蛋白相互作用数据库HPRD (http://www.) 的数据来寻找他的DEGs (differentially expressed genes) 的相互作用关系,然后使用Cytoscape 进行构建网络图。
做出了三个网络图,分为下调基因的网络图,上调基因的网络图,总差异表达基因的网络图。并且只表示第一层相互作用的蛋白。单一节点和自己和自己相互作用的蛋白被移除。结果图类似是这样的:

由于敲除的基因是Ezrin,其蛋白EZR作为query node与其他DEGs的相互作用网络也被做了出来:

其次是网络拓普结构分析。
使用NetworkAnalyzer进行拓普分析。由于如果某个网络节点度满足幂律分布则表示这个网络属于无尺度网络。PPI网络也遵循这一定律。为了验证上面的三个ppi网络是否是无尺度网络,作者做了三个网络(下调的、上调的和总体的)的节点度分布:

发现这三个网络都是满足幂律分布的,这就表明PPI网络是无尺度的。为什么要证明这个呢,其实这个和WGCNA分析的原理是一样的,出发点就是在一个调控网络中起关键作用的节点是很少的,但存在这样的起关键作用的节点。然后进行寻找这样的hubs (中心枢纽) 。
作者不光在HPRD中寻找蛋白相互作用关系,还使用了STRING数据库的(这个库里不光有实验验证的,还有生信预测的相互作用结果)。然后再作节点度分析,发现使用两个数据库都能得到相似的结果,下面是STRING数据库的度分布结果:

使用两个数据库的数据来验证,表明他们的DEGs PPI网络是可靠和稳定的。
接下来是PPI网络中蛋白的亚细胞定位。
蛋白亚细胞定位的数据是从Uniprot中获得的。然后每个蛋白的亚细胞定位数据导入Cytoscape作为一列,使用Cytoscape的插件Cerebral (http://www./cerebral/) 即可构建基于亚细胞定位的PPI网络,如下图:

作者还做了只与EZR 相互作用的蛋白的亚细胞定位PPI:

再将相互作用深度加深,获得与EZR相互作用、间接相互作用蛋白的相互作用网络:

并且在之前的研究中作者发现Ezrin敲除后上调ATF3,然后他就寻找EZR到ATF3的最短路径,发现了9个路劲:


由于大部分信号转导途径是从基质到细胞核,所以根据上图的亚细胞定位PPI的结果,就可以给出最有可能的四个途径:
EZR→ACTB→SMAD3→ATF3;
EZR→PRKAR2A→ SMAD3→ATF3;
EZR→CTNNB1→NFKB1→ATF3;
EZR→ WWOX→TP53→ATF3.
接下来构建功能注释网络图:
作者使用ClueGo插件来构建BP (Biological process) 网络。并且设定kappa score 阈值为0.3. 结果是这样的:

最后使用随机游走方法来寻找重要的DEGs.
通过有重启的随机游走(RWR)获得与EZR 关联最紧密的DEGs。然后获得了每个DEG的可能性得分。得分越高表示蛋白与EZR关联越紧密。然后将得分导入Cytoscape作为列属性作图,点越大得分越高:

只显示DEGs的RWR分析的PPI网络: